組蛋白修飾:基因調控和疾病發病機制的意義
日期:2024-02-06 11:36:44
組蛋白修飾是表觀遺傳學中一個迷人而復雜的領域,它研究染色質結構的動態變化。這一非凡的過程通過組蛋白的化學變化對基因表達、轉錄調控和各種細胞功能產生深遠影響。通過組蛋白標記的書寫者、擦除者和閱讀者之間復雜的相互作用,組蛋白密碼成為一種極具吸引力的表觀遺傳調控語言。
本文探討了組蛋白修飾的性質、功能意義、機制、串擾、動態調控以及對疾病的影響。
1. 什么是組蛋白修飾?
組蛋白修飾是指發生在組蛋白上的化學變化,包括在組蛋白尾部的特定氨基酸殘基上添加各種化學基團,如乙酰基、甲基、磷酸基、泛素等。這些修飾不會改變 DNA 核苷酸序列,但可以調節 DNA 對轉錄機制的可及性。
2. 組蛋白修飾的功能
組蛋白修飾在基因調控和染色質結構中發揮著多種多樣的重要功能。組蛋白修飾作為一種調控代碼,有助于微調基因表達模式、維持基因組穩定性,以及在發育過程中協調細胞過程并對環境線索做出反應。
2.1 基因激活或抑制
組蛋白修飾可松弛染色質結構,使 DNA 更容易被轉錄機制利用,從而促進基因激活。組蛋白上特定賴氨酸殘基的乙酰化可中和其正電荷,降低組蛋白與 DNA 之間的親和力,從而提高轉錄活性。某些賴氨酸或精氨酸殘基的甲基化也能通過創造一個有利的染色質環境來增強基因表達。
組蛋白修飾也可抑制基因表達,使染色質更加緊湊,轉錄沉默。特定賴氨酸殘基(如 H3K9 和 H3K27)的甲基化與基因抑制有關。這些修飾會招募蛋白與修飾的組蛋白結合,誘導染色質壓實,抑制轉錄因子和其他調控蛋白與 DNA 的結合。
2.2 染色質重塑
組蛋白修飾在染色質結構的動態重塑過程中起著至關重要的作用 [1]。它們是染色質重塑復合物招募的信號,可改變核小體沿 DNA 的定位和組織。這一重塑過程可改變基因的可及性,促進轉錄復合物在特定基因啟動子或增強子上的組裝。
2.3 表觀遺傳記憶
組蛋白修飾有助于表觀遺傳記憶的建立和維持。某些修飾可通過細胞分裂甚至跨代遺傳,有助于基因表達模式的穩定維持。例如,在發育過程中,特定的組蛋白修飾可將基因標記為激活或抑制,確保其在不同類型細胞中的適當表達模式。
2.4 DNA 修復和復制
組蛋白修飾參與 DNA 修復和復制過程的調控。它們招募負責維護基因組完整性和協調 DNA 修復機制的蛋白質和復合體。此外,組蛋白修飾還通過組織染色質結構和促進復制機器沿 DNA 鏈移動,在確保 DNA 正常復制方面發揮作用。
2.5 發育過程
組蛋白修飾是胚胎發生、組織分化和細胞命運決定等各種發育過程不可或缺的組成部分。它們有助于建立特定的基因表達程序,從而指導細胞分化,確保不同類型細胞和組織的適當發育。
2.6 環境反應
組蛋白修飾可根據環境線索和刺激進行動態調節。壓力、營養和暴露于毒素等外部因素可誘導組蛋白修飾發生變化,從而導致基因表達模式和細胞反應的改變。
3. 常見的組蛋白修飾及其機制
迄今為止,已發現和研究的組蛋白修飾類型眾多。其中最著名和研究最廣泛的組蛋白修飾包括乙酰化、甲基化、磷酸化、泛素化、蘇木酰化和 ADP 核糖基化。這些修飾在特定殘基上的不同組合和水平,造就了組蛋白修飾功能的多樣性和復雜性。
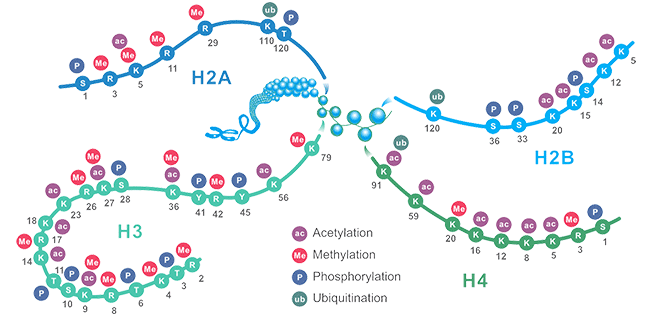
圖1. 常見組蛋白修飾位點
3.1 組蛋白乙酰化
組蛋白乙酰化是指乙酰輔酶 A 的乙酰基(COCH3)轉移到組蛋白賴氨酸殘基上的過程。組蛋白乙酰轉移酶(HAT)和去乙酰化酶(HDAC)是負責寫入和清除組蛋白尾部乙酰化的酶 [2]。組蛋白 H3 和 H4 中的賴氨酸殘基是 HAT 復合物的首選目標。
這種組蛋白修飾與基因激活有關,因為它中和了賴氨酸的正電荷,導致染色質結構更加松弛,轉錄活性增強。它密切參與許多細胞過程的調控,包括染色質動力學和轉錄、基因沉默、細胞周期進展、細胞凋亡、分化、DNA 復制、DNA 修復、核導入和神經元抑制。
下表列出了部分可檢測不同乙酰化組蛋白的 CUSABIO 乙酰化組蛋白抗體。
表1. CUSABIO 乙酰化組蛋白抗體
| Target | Product Name | Code | Tested Species | Tested Applications |
|---|---|---|---|---|
| H2AFZ | Acetyl-H2AFZ (K11) Antibody | CSB-PA010100PA11acHU | Human | ELISA, WB, IF, ChIP |
| HIST1H1B | Acetyl-HIST1H1B (K16) Antibody | CSB-PA010377PA16acHU | Human | ELISA, IF, ChIP |
| HIST1H1C | Acetyl-HIST1H1C (K16) Antibody | CSB-PA010378PA16acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H1C | Acetyl-HIST1H1C (K62) Antibody | CSB-PA010378PA62acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H1C | Acetyl-HIST1H1C (K74) Antibody | CSB-PA010378OA74acHU | Human | ELISA, IF, ChIP |
| HIST1H1C | Acetyl-HIST1H1C (K84) Antibody | CSB-PA010378OA84acHU | Human | ELISA, WB, ICC, ChIP |
| HIST1H1E | Acetyl-HIST1H1E (K16) Antibody | CSB-PA010380PA16acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H1E | Acetyl-HIST1H1E (K33) Antibody | CSB-PA010380PA33acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H1E | Acetyl-HIST1H1E (K51) Antibody | CSB-PA010380PA51acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H1E | Acetyl-HIST1H1E (K63) Antibody | CSB-PA010380PA63acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H2AG | Acetyl-HIST1H2AG (K36) Antibody | CSB-PA010389PA36acHU | Human | ELISA, WB, ICC, IF, ChIP |
| HIST1H2BB | Acetyl-HIST1H2BB (K16) Antibody | CSB-PA010402OA16acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H2BB | Acetyl-HIST1H2BB (K5) Antibody | CSB-PA010402NA05acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H2BC | Acetyl-HIST1H2BC (K108) Antibody | CSB-PA010403OA108acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H2BC | Acetyl-HIST1H2BC (K116) Antibody | CSB-PA010403OA116acHU | Human, Rat | ELISA, WB, IF, ChIP |
| HIST1H2BC | acetyl-Histone H2B (K120) Antibody | CSB-PA010403OA120acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H3A | Acetyl-HIST1H3A (K36) Antibody | CSB-PA010418OA36acHU | Human, Rat | ELISA, WB, IF, ChIP |
| HIST1H3A | Acetyl-HIST1H3A (K37) Antibody | CSB-PA010418OA37acHU | Human, Rat | ELISA, WB, IF, ChIP |
| HIST1H3A | Acetyl-HIST1H3A (K79) Antibody | CSB-PA010418OA79acHU | Human | ELISA, IF, ChIP |
| HIST1H3A | Acetyl-HIST1H3A (K9) Antibody | CSB-PA010418PA09acHU | Human | ELISA, WB, ICC, IF, ChIP |
| HIST1H3A | Acetyl-HIST1H3A (T22) Antibody | CSB-PA010418OA22acHU | Human | ELISA, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K12) Antibody | CSB-PA010429PA12acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K12) Antibody | CSB-PA010429OA12acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K16) Antibody | CSB-PA010429NA16acHU | Human | ELISA, WB, ICC, IF, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K16) Antibody | CSB-PA010429PA16acHU | Human | ELISA, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K31) Antibody | CSB-PA010429OA31acHU | Human, Rat | ELISA, WB, ICC, IF, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K5) Antibody | CSB-PA010429PA05acHU | Human | ELISA, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K79) Antibody | CSB-PA010429OA79acHU | Human | ELISA, ICC, IF, ChIP |
| HIST1H4A | Acetyl-HIST1H4A (K8) Antibody | CSB-PA010429PA08acHU | Human | ELISA, ICC, IF, ChIP |
3.2 組蛋白甲基化
組蛋白甲基化特異性地發生在組蛋白 H3 和 H4 的不同賴氨酸或精氨酸殘基上。賴氨酸可以是單甲基化、雙甲基化或三甲基化,精氨酸可以是單甲基化或雙甲基化(不對稱或對稱)[3,4]。它會導致轉錄抑制或激活,具體取決于被甲基化的特定尾部殘基及其甲基化程度。例如,組蛋白 H3 在賴氨酸 4(H3K4)、H3K36、H3K79 或精氨酸 17(H3R17)處的甲基化與轉錄激活有關 [5]。相反,組蛋白 H3 在 H3K9、H3K27 或組蛋白 H4 在 H4K20 處的甲基化通常與轉錄抑制有關。
組蛋白甲基化通過招募各種轉錄因子影響基因表達,而不是直接改變染色質結構 [6]。
下表列出了部分可檢測不同甲基化組蛋白的 CUSABIO 甲基化組蛋白抗體。
表2. CUSABIO 甲基化組蛋白抗體
| Target | Product Name | Code | Tested Species | Tested Applications |
|---|---|---|---|---|
| H1F0 | Mono-methyl-H1F0 (K101) Antibody | CSB-PA010087OA101me1HU | Human, Rat | ELISA, WB, ICC, IF, ChIP |
| H1F0 | Mono-methyl-H1F0 (K81) Antibody | CSB-PA010087OA81me1HU | Human | ELISA, ICC, IF, ChIP |
| H2AFZ | Mono-methyl-H2AFZ (K4) Antibody | CSB-PA010100OA04me1HU | Human | ELISA, ICC, IF, ChIP |
| H3F3A | Di-methyl-H3F3A (K79) Antibody | CSB-PA010109OA79me2HU | Human | ELISA, WB, ChIP |
| HIST1H1C | Mono-methyl-HIST1H1C (K118) Antibody | CSB-PA010378PA118me1HU | Human | ELISA, IF, ChIP |
| HIST1H1C | Mono-methyl-HIST1H1C (K186) Antibody | CSB-PA010378PA186me1HU | Human, Mouse, Rat | ELISA, WB, IF, ChIP |
| HIST1H1C | Di-methyl-HIST1H1C (K45) Antibody | CSB-PA010378OA45me2HU | Human | ELISA, WB, ICC, IP, ChIP |
| HIST1H1C | Mono-methyl-HIST1H1C (K96) Antibody | CSB-PA010378OA96me1HU | Human, Mouse, Rat | ELISA, WB, IF, ChIP |
| HIST1H1E | Di-methyl-HIST1H1E (K16) Antibody | CSB-PA010380PA16me2HU | Human | ELISA, ICC, IF, ChIP |
| HIST1H2AG | Mono-methyl-HIST1H2AG (K9) Antibody | CSB-PA010389OA09me1HU | Human | ELISA, ICC, IF, ChIP |
| HIST1H2AG | Di-methyl-HIST1H2AG (R29) Antibody | CSB-PA010389OA29me2HU | Human | ELISA, ChIP |
| HIST1H2BC | Mono-methyl-HIST1H2BC (K12) Antibody | CSB-PA010403OA12me1HU | Human, Mouse, Rat | ELISA, WB, ICC, ChIP |
| HIST1H2BC | Mono-methyl-HIST1H2BC (K20) Antibody | CSB-PA010403OA20me1HU | Human | ELISA, WB, ChIP |
| HIST1H4A | Mono-methyl-HIST1H4A (K5) Antibody | CSB-PA010429OA05me1HU | Human | ELISA, IF, ChIP |
3.3 組蛋白磷酸化
組蛋白磷酸化是指在組蛋白尾部的絲氨酸、蘇氨酸或酪氨酸殘基上添加磷酸基團。它與信號通路有關,在染色質重塑、轉錄激活以及細胞周期和 DNA 損傷反應等細胞過程中發揮作用。
組蛋白磷酸化由兩種作用截然不同的酶調節。激酶增加磷酸基團,而磷酸酶則去除磷酸基團。磷酸化組蛋白具有多種功能,包括 DNA 損傷修復、有絲分裂和減數分裂過程中染色質壓實的調節、細胞凋亡以及轉錄活性的調節 [7,8]。組蛋白磷酸化最著名的功能發生在細胞對 DNA 損傷的反應過程中,磷酸化的組蛋白 H2A(X)會在 DNA 斷裂位點周圍劃分出大的染色質結構域 [9]。
下表列出了部分可檢測不同磷酸化組蛋白的 CUSABIO 磷酸化組蛋白抗體。
表3. CUSABIO 磷酸化組蛋白抗體列表
| Target | Product Name | Code | Tested Species | Tested Applications |
|---|---|---|---|---|
| H2AFX | Phospho-H2AFX (S139) Antibody | CSB-PA010097OA139phHU | Human | ELISA, WB, IF, ChIP |
| HIST1H1B | Phospho-HIST1H1B (T154) Antibody | CSB-PA010377PA154phHU | Human | ELISA, WB, IF, ChIP |
| HIST1H1D | Phospho-HIST1H1D (T179) Antibody | CSB-PA010379OA179phHU | Human | ELISA, IF, ChIP |
| HIST1H3A | Phospho-HIST1H3A (S28) Antibody | CSB-PA010418OA28phHU | Human | ELISA, IF, ChIP |
| HIST1H3A | Phospho-HIST1H3A (T6) Antibody | CSB-PA010418OA06phHU | Human | ELISA, IF, ChIP |
| HIST1H3A | Phospho-HIST1H3A (T80) Antibody | CSB-PA010418OA80phHU | Human | ELISA, IF, ChIP |
3.4 組蛋白泛素化
組蛋白可通過添加泛素對賴氨酸殘基進行廣泛修飾,泛素的添加受一系列酶的調控,包括 E1 激活酶、E2 連接酶和 E3 連接酶。相反,去泛素化酶控制著泛素從組蛋白中的清除。組蛋白泛素化的這種動態調控在維持基因組穩定性、調節細胞周期和調控轉錄過程方面發揮著至關重要的作用。
3.5 組蛋白總酰化
組蛋白蘇木酰化涉及組蛋白上特定賴氨酸殘基與小泛素樣修飾劑(SUMO)基團的共價連接。Sumoylation 可發生在各種組蛋白上,包括組蛋白 H2A、H2B、H3 和 H4 及其變體。與泛素化類似,SUMOylation 也是一個多步驟過程,由包括 SUMO 激活酶(E1)、SUMO 結合酶(E2)和 SUMO 連接酶(E3)在內的多種酶的連續作用介導。
組蛋白蘇木酰化通過抑制某些高階染色質結構的形成,在調節染色質結構方面發揮著關鍵作用。它是招募基因激活和抑制因子的關鍵信號。
3.6 組蛋白 ADP-核糖基化
組蛋白 ADP 核糖基化是指在組蛋白尾部的谷氨酸、天冬氨酸和精氨酸等多種氨基酸殘基上添加 ADP 核糖分子。ADP-ribosylation 由 ADP-ribosyltransferases(ARTs)催化,是一個受 ADP-ribosyl hydrolases 調節的可逆過程。
4. 不同組蛋白修飾之間的相互影響
2000 年,Strahl 和 Allis 提出了 "組蛋白密碼",它是由單個或多個組蛋白修飾之間的相互作用形成的[10]。組蛋白修飾通常會相互串擾,影響彼此的作用。不同組蛋白修飾之間的串擾為染色質結構和基因表達的調控增加了另一層復雜性。
例如,組蛋白 H3 上絲氨酸 10 的磷酸化(H3S10ph)會阻礙組蛋白 H3 上賴氨酸 9 的三甲基化(H3K9me3),同時促進組蛋白 H4 上賴氨酸 16 的乙酰化(H4K16ac)[11]。
Walter 等人在芽殖酵母中發現,Snf1 激酶磷酸化組蛋白 H3 上的絲氨酸 10(S10),加速了 Gcn5 乙酰轉移酶對 H3 賴氨酸 14(K14)的乙酰化,從而增加了基因激活過程中 H3 與 14-3-3 蛋白 Bmh1 和 Bmh2 的相互作用 [12]。
Daujat 等人發現,在哺乳動物細胞中,H3 在 K18 和 K23 上的乙酰化會刺激 CARM1 甲基轉移酶對精氨酸 17(R17)的甲基化,從而導致雌激素反應基因的活化 [13]。
此外,組蛋白修飾串聯也會影響特定修飾的缺失。例如,在芽殖酵母中,Lee 和 Shilatifard 證實 RNA 聚合酶 II 相關的 Set2 甲基轉移酶會甲基化 H3K36,從而建立一個標記,引導核小體在 RNA 聚合酶通過后由 Rpd3S 去乙酰化酶復合物進行 H3 和 H4 去乙酰化 [14]。
5. 組蛋白修飾的動態調控
組蛋白修飾由被稱為 "書寫者"、"擦除者 "和 "閱讀者 "的酶或蛋白質的復雜相互作用進行動態調控。寫入器通過酶來修飾組蛋白上的特定氨基酸殘基,包括 HATs、HMTs/KMTs、PRMTs、激酶和泛素連接酶。清除劑可去除或逆轉這些修飾,包括 HDAC、HDM/KDM、磷酸酶和 DUB。包括溴化結構域、染色質結構域或都鐸結構域在內的閱讀器可以識別和鑒定這些修飾的組蛋白殘基,然后將這些信號轉化為不同的下游效應。例如,溴結構域可選擇性地靶向乙酰賴氨酸殘基。許多染色質結合甲基化的賴氨酸,而 Tudor 結構域則結合甲基化的精氨酸。組蛋白修飾與調控蛋白之間的相互作用影響著染色質結構、基因表達和細胞過程。
6. 組蛋白修飾在疾病中的作用
組蛋白修飾的激活或失活失衡會擾亂基因表達程序,并導致與轉錄組異常相關的發病機制。因此,與組蛋白修飾相關的基因突變可能會導致癌癥、自身免疫性疾病、炎癥性疾病和神經退行性疾病等各種疾病的發生和發展。
異常的組蛋白修飾,如增加的組蛋白乙酰化或改變的組蛋白甲基化模式,可導致腫瘤發生。一項研究表明,在各種癌癥類型中,H4K16ac 和 H4K20me3 的消耗,確定了這兩種組蛋白標記是腫瘤的明顯特征,以及 H4K16ac 與腫瘤進展的相關性 [15,16]。Seligson DB 等人發現,H3K18ac、H4K12ac 和 H4R3me2 與前列腺癌分級呈正相關 [17]。
表觀遺傳失調,包括組蛋白修飾的改變,與類風濕性關節炎 [18]和系統性紅斑狼瘡 [19]等自身免疫性疾病有關。組蛋白乙酰化、甲基化和磷酸化的變化會影響免疫細胞的功能、細胞因子的產生和免疫反應的調節,從而導致自身免疫性疾病的發生。
組蛋白修飾與阿爾茨海默病、帕金森病和亨廷頓病等神經退行性疾病有關 [20,21]。組蛋白乙酰化、甲基化和磷酸化模式的改變與神經炎癥、神經細胞死亡和突觸可塑性受損有關,而這些正是這些疾病的標志性特征。
組蛋白修飾可通過調節心臟細胞和血管組織中的基因表達模式影響心血管健康。組蛋白乙酰化和甲基化失調與心臟肥大、心力衰竭、動脈粥樣硬化和血管重塑有關。
7. 結論
組蛋白修飾是調控基因表達和染色質結構的重要表觀遺傳標記。這些修飾就像一種動態語言,形成了組蛋白密碼,調控著包括發育、分化和疾病進展在內的各種細胞過程。了解它們的性質、功能意義、作用機制、相互影響、動態調控以及對疾病的影響,將為治療干預開辟新的途徑,并加深我們對組蛋白修飾在健康和疾病中的了解。
參考文獻:
[1] Bannister, A. J. & Kouzarides, T. Regulation of chromatin by histone modifications [J]. Cell Res. 21, 381–395 (2011).
[2] Marmorstein, R. & Zhou, M. M. Writers and readers of histone acetylation: structure, mechanism, and inhibition [J]. Cold Spring Harb. Perspect. Biol. 6, a018762 (2014).
[3] Bannister, A. J., Schneider, R., and Kouzarides, T. (2002). Histone methylation [J]. Cell 109, 801–806.
[4] Bannister, A. J., and Kouzarides, T. (2005). Reversing histone methylation [J]. Nature 436, 1103–1106.
[5] Bauer, U. M., Daujat, S., et al. Methylation at arginine 17 of histone H3 is linked to gene activation [J]. EMBO Rep. 3, 39–44 (2002).
[6] Klose, R. J., and Zhang, Y. (2007). Regulation of histone methylation by demethylimination and demethylation [J]. Nat. Rev. Mol. Cell Biol. 8, 307–318.
[7] Rossetto D, Avvakumov N, C?té J. Histone phosphorylation: a chromatin modification involved in diverse nuclear events [J]. Epigenetics. 2012;7:1098–108.
[8] Bannister AJ, Kouzarides T. Regulation of chromatin by histone modifications [J]. Cell Res. 2011;21:381–95.
[9] Ye Zhang, Karen Griffin, et al. Phosphorylation of Histone H2A Inhibits Transcription on Chromatin Templates [J]. J Biol Chem. 2004, 279(21):21866-72.
[10] Latham JA, Dent SY. Cross-regulation of histone modifications [J]. Nat Struct Mol Biol. 2007;14:1017–24.
[11] Zippo, A.; Serafini, R.; et al. Histone Crosstalk between H3S10ph and H4K16ac Generates a Histone Code That Mediates Transcription Elongation [J]. Cell 2009, 138, 1122–1136.
[12] Walter W, Clynes D, et al. 14-3-3 interaction with histone H3 involves a dual modification pattern of phosphoacetylation [J]. Mol Cell Biol. 2008 Apr;28(8):2840-9.
[13] Sylvain Daujat, Uta-Maria Bauer, et al. Crosstalk between CARM1 methylation and CBP acetylation on histone H3 [J]. Curr Biol. 2002 Dec 23;12(24):2090-7.
[14] Jung-Shin Lee and Ali Shilatifard. A site to remember: H3K36 methylation a mark for histone deacetylation [J]. Mutat Res. 2007 May 1;618(1-2):130-4.
[15] Fraga MF, Ballestar E, et al. Loss of acetylation at Lys16 and trimethylation at Lys20 of histone H4 is a common hallmark of human cancer [J]. Nat Genet. 2005;37:391-400.
[16] Tryndyak VP, Kovalchuk O, Pogribny IP. Loss of DNA methylation and histone H4 lysine 20 trimethylation in human breast cancer cells is associated with aberrant expression of DNA methyltransferase 1, Suv4-20h2 histone methyltransferase and methyl-binding proteins [J]. Cancer Biol Ther. 2006;5:65-70.
[17] Seligson DB, Horvath S, et al. Global histone modification patterns predict risk of prostate cancer recurrence [J]. Nature. 2005;435:1262-1266.
[18] Araki, Y. et al. Histone methylation and STAT-3 differentially regulate interleukin-6-induced matrix metalloproteinase gene activation in rheumatoid arthritis synovial fibroblasts [J]. Arthritis Rheumatol. 68, 1111–1123 (2016).
[19] Hu N, Qiu X, Luo Y, Yuan J, Li Y, Lei W, et al. Abnormal histone modification patterns in lupus CD4+ T cells [J]. J Rheumatol (2008) 35:804–10.
[20] Santana DA, Smith MAC, Chen ES. Histone Modifications in Alzheimer's Disease [J]. Genes (Basel). 2023 Jan 29;14(2):347.
[21] Park, J., Lee, K., Kim, K. et al. The role of histone modifications: from neurodevelopment to neurodiseases. Sig Transduct Target Ther 7, 217 (2022).
上一篇: 組蛋白去甲基化酶:LSD和JmjC家族
下一篇: 組蛋白修飾如何影響基因表達?










